· Wissenschaftliche Publikationen
Alexandra Pitt, Stefan Lienbacher, Johanna Schmidt, Meina Neumann‑Schaal, Jacqueline Wolf, and Martin W. Hahn (2024). Description of a new freshwater bacterium Aquirufa regiilacus sp. nov., classification of the genera Aquirufa, Arundinibacter, Sandaracinomonas, and Tellurirhabdus to the family Spirosomataceae, classification of the genus Chryseotalea to the family Fulvivirgaceae and Litoribacter to the family Cyclobacteriaceae, as well as classification of Litoribacter alkaliphilus as a later heterotypic synonym of Litoribacter ruber. Archives of Microbiology, Volume 206, article number 79. https://link.springer.com/article/10.1007/s00203-023-03801-8
· Im Projekt erhaltene Daten
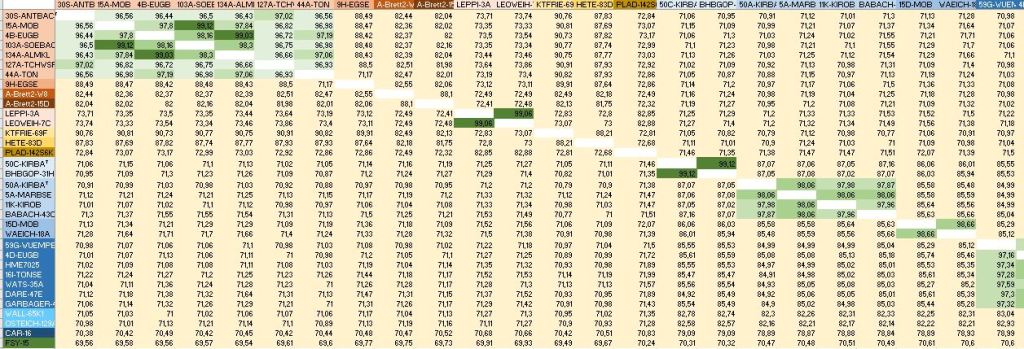
In dieser Excel-Tablelle finden sich alle Daten zu den im Projekt genommenen Wasserproben. Dank der Mitarbeit vieler Citizen Scientists konnten fast 180 Wasserproben in die Kultivierung von Bakterienstämmen einbezogen werden. In der Tabelle sind die 58 Wasserproben, aus denen Aquirufa-Kulturen gewonnen werden konnten, rot markiert.
· In öffentlichen Datenbanken verfügbare Daten
Datenbank des National Centers of Biotechnology Information (USA)
Hier sind Daten zu einigen mit Hilfe der Citizen Scientists im Projekt isolierten Bakterienstämmen öffentlich verfügbar. Beim betreffenden Stamm ist der Name des Cititzen Scientist, von dem Wasserprobe und Vorkulturen stammen, angegeben.
PRJNA929697 Aquirufa strains, genome sequencing and assembly, Isolation and genome sequencing of Aquirufa strains within a Citizen Science Project
SAMN32969352: LEPPI-3A, Ioanna Polo
SAMN32970148: WAEICH-18A, Martin Windberger
SAMN32970375: BABACH-43C, Martha Wohlschlaeger
SAMN32970536: BHBGOP-31H, Hannah Stockinger
SAMN32971549: DARE-47E, Sonia Qaiser
SAMN32971568: LEOWEIH-7C, Edna Jahija
SAMN32971698: WATS-35A, Kilian Ponget
SAMN32972266: GARBAGER-33B, Julia Schiller
SAMN38274655: HETE-83D, Katharina Strohbichler
SAMN38274673: KTFRIE-69F, Matthias Reiterer
SAMN38274714: OSTEICH-129A, David Eder
SAMN38274715: PLAD-142S6K, Lena Mayrhofer
SAMN38691316: OSTEICH-129V, David Eder
SAMN38691364: 1-SAACH-A3, Lucie Meznerová, Zoe Huber, Anna Stephens
SAMN38879877: INPA-49A, Sophia Lindorfer
SAMN38879952: DOPA-48B, Sophia, Lindorfer
SAMN38880992: PLAD-142H, Lena Mayrhofer
SAMN38881031: FRTEICH-102D4, Xaver Golmayer
SAMN39327898: ROBBACH-80M, Alexandra Brkovic
SAMN39327919: VOEWA-120C1, Lina Sophie Ematinger
SAMN39327938: SAAGRA-154A, Sarah Koeckerbauer
SAMN39327896 : HALL-GUE30-B3, Florian Wolff
SAMN40100718: 2-AUSEE-172A13, Armin Omercevic
SAMN40100712: 2-AUSEE-184A6, Mexxi Roenfeld
SAMN40891753: 2-BAHN-186B, Miriam Fenninger
Datenbank Integrated Microbial Genomes & Microbiomes
Von den in der obigen Liste enthaltenen Bakterienstämmen haben wir das gesamte Genom -also die gesamte Erbsubstanz- sequenzieren lassen. In der Datenbank sind diese Genomsequenzen sowie die sogenannten Annotierungen enthalten. Das bedeutet, dass man hier sehen kann, welche Gene die einzelnen Bakterienstämme besitzen und was noch viel wichtiger ist, welche Funktionen diese Gene haben. Man kann daraus Informationen zur Lebensweise und Ökologie der Bakterien ableiten.